Spectral Clustering - 算法解析與 numpy 程式實作
前言
在社群網絡中分析中,有時候會想要找出網絡當中的社群(community detection),一個最直觀的想法是直接對網絡進行分群,那麼分群結果就是各個community了。
如果我們能夠計算各個節點之間的相似性,那麼我們當然可以直接套用傳統的分群方法,如cosine similiarity,但一來是相似度特徵可能很難取得,二來是這樣分群的話就沒有利用到網絡的結構了,這時我們可以改為使用基於圖論的分群方法。
今天這篇文章會帶大家簡單瞭解什麼是cut approach, balanced-cut approach以及其代表方法spectral clustering,並示範不依賴其他套件,僅使用numpy實作spectral clustering。
大綱
內文
Mini-cut Approach
Cut approach,顧名思義是將網絡給切開來形成多個網絡,屬於partitional clustering的一種,每個節點只會屬於一個community,並且community之間也不會overlapping,而Mini-cut Approach則是希望在切割網絡時能最小化切掉的edge數量。
聽起來很直觀,但這樣切會有一個問題,那就是有可能會切出一些非常小的community,導致分群不夠balance。
Balanced-cut Approach
為了解決Mini-cut Approach的缺點,Balanced-cut Approach在切割網絡時除了考量原本的edge,另外除以community的node數做加權調整,切割的目標從最小化edge數轉為最小化ratio cut,公式如下:
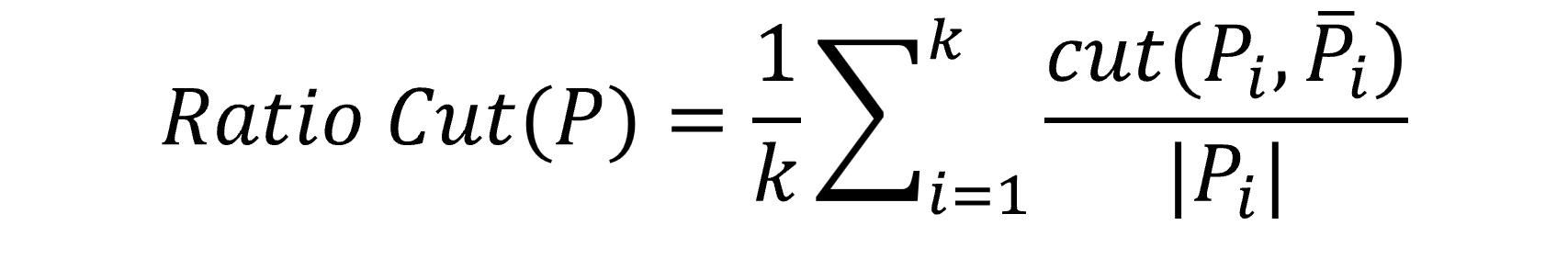
公式解釋: 分成k群(k要自己決定),每次取一個群Pi出來看,分子部分表示將Pi切開的話會切到的edge數,分母部分表示Pi內部的node數,將k個群的結果加總起來就會是整個網路的ratio cut分數。
Matrixs
為了要計算Ratio Cut,我們先要來認識一下三種矩陣:
- Diagonal Degree Matrix 度數矩陣
Degree(度數)指的是一個node有多少個鄰居,將這些Degree轉換為對角矩陣(Diagonal Matrix)就得到Diagonal Degree Matrix。
1 | D = np.array([ |
- Adjacency Matrix 鄰接矩陣
只要是對Graph有些概念的人應該都對Adjacency Matrix不太陌生,Adjacency Matrix描述了node之間的關係,下面例子假設node之間的關係都是雙向的,所以會是個對稱矩陣。
1 | A = np.array([ |
- Laplacian Matrix 調和矩陣(拉普拉斯矩陣)
Laplacian Matrix 的算法就是直接將Diagonal Degree Matrix 減去 Adjacency Matrix,聽起來可能讓人滿頭問號,為什麼要這麼做? 這邊先賣個關子,繼續看下去就知道了。
1 | L = np.array([ |
Calculation
現在先假設我們已經知道分群的結果了,並將結果以0,1的形式表示為一個matrix,令這個結果為X,下面結果是我隨便分的,分3群,X[i][j]==1表示nodei屬於clusterj。1
2
3
4
5
6
7
8X = np.array([
[1, 0, 0],
[1, 0, 0],
[0, 0, 1],
[0, 0, 1],
[0, 1, 0],
[0, 1, 0],
[0, 1, 0]])
- Diagonal Degree Matrix 解釋
我們先將X的轉置矩陣與D做內積(X.T.dot(D)):
1 | array([[2, 4, 0, 0, 0, 0, 0], |
觀察一下結果會發現,這個矩陣就只是是把Degree填到對應的cluster中而已。
接著我們再把上面結果與X做內積(X.T.dot(D).dot(X)):1
2
3array([[6, 0, 0],
[0, 7, 0],
[0, 0, 5]])
這個結果其實就表示每個cluster的degree總和。
- Adjacency Matrix 解釋
接著我們直接將上面公式的D換成A(X.T.dot(A).dot(X)):
1 | array([[2, 2, 2], |
會發現結果表示在cluster與cluster間互相連接的edge數。
- Laplacian Matrix 解釋
如果我們將兩個結果相減的話,會發現每一行的第i個元素(對角)就是我們想要計算的cut數(分子部分)!!!
1 | array([[ 4, -2, -2], # i=0, 4 |
接著我們另外計算每個cluster裡面共有多少個node,我們可以將X的轉置後與自己相乘(X.T.dot(X)):1
2
3array([[2, 0, 0],
[0, 3, 0],
[0, 0, 2]])
最後將對角的結果相除後加起來就是算平均就得到Ratio Cut的分數啦~~~
(我這邊直接矩陣相除,大家可以自己算。)1
2
3array([[ 2. , -inf, -inf],
[-inf, 1. , -inf],
[-inf, -inf, 1.5]])
根據上面矩陣運算的過程,我們能將公式重新整理為:
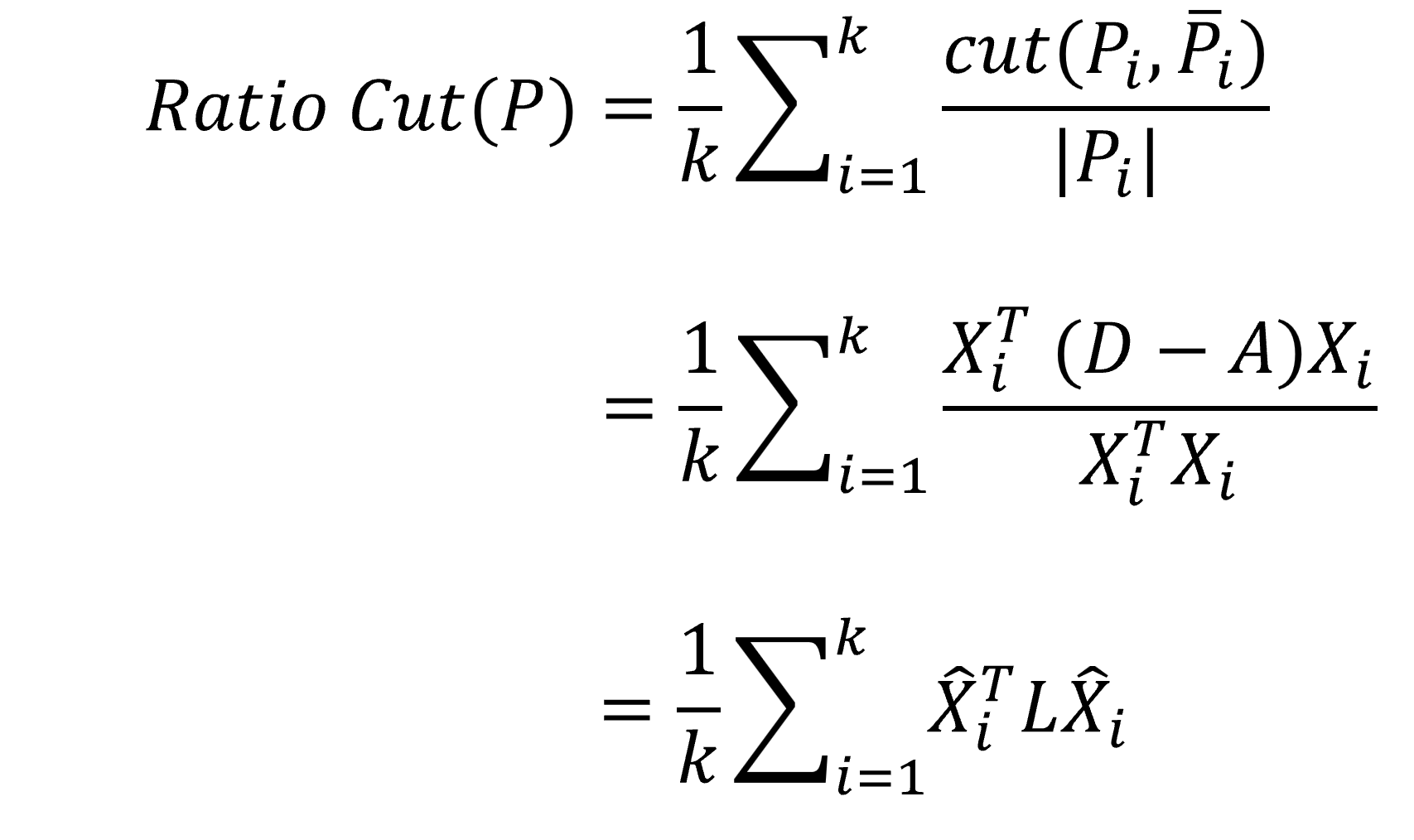
在第三行的部分,我們另外對X進行標準化(同除以分母)變成了X prime。
Minimize
回到最一開始的問題,我們的目標是要最小化ratio cut的分數,方法就是透過上面的公式去求解X prime矩陣,難過的是,還記的X矩陣裡最一開始只能填入0跟1嗎,想要在這樣嚴格的限制下求出最佳解是個NP-hard的問題。
不過不要灰心,我們還是可以放寬條件,套用近似演算法,其中一種方法就是接下來要介紹的Spectral Clustering~~~
Spectral Clustering
Spectral Clustering 基於ratio cut,不過X矩陣中可以填入的值變成介於0~1之間,在放寬條件後,這個最小化問題的解可以被證明是求解Laplacian Matrix的前k小個特徵值的特徵向量。
- Eigenvalue & Eigenvactors
我們可以用np.linalg.eig來替我們計算特徵值與特徵向量,並使用np.argsort來替我們找出最小的k個特徵向量。
1 | # calculate eigenvalue and eigenvector |
得到的結果如下:1
2
3
4
5
6
7array([[ 0.37796447, -0.45244521, -0.41243936],
[ 0.37796447, 0.17351892, -0.08541899],
[ 0.37796447, 0.54280925, -0.41243936],
[ 0.37796447, 0.32598548, -0.08541899],
[ 0.37796447, -0.58986844, -0.08541899],
[ 0.37796447, -0.09036404, 0.37705765],
[ 0.37796447, 0.09036404, 0.70407803]])
- K-means
由於得到的特徵值並無法直接拿來做解釋,因此Spectral Clustering另外使用了K-means來為這些特徵值做分群,因為最小的特徵值為0、特徵向量一定是常數,所以我們可以把它刪去,也就是說,如果我們想要分成k群,那我們至少需要使用k-1個特徵向量。
下面程式提供一個簡單的K-means範例,可以根據自己的需求另外設置停止條件。
1 | def k_means(X, k, max_iters=10000): |
這樣就得到分群結果啦~~~(注意: 實際執行數字的順序可能不一樣)1
array([2, 0, 0, 0, 2, 1, 1], dtype=int64)
結語
其實忽略到複雜的數學證明的話,Spectral Clustering的概念還挺直觀的,文章參考了台大資管系的陳建錦教授所使用的上課講義,經過本人吸收轉化撰寫而成。